English Version : https://www.linkedin.com/pulse/sql-server-machine-learning-services-wine-quality-romain-casteres/
Dans cet article je vais présenter SQL Server Machine Learning services, nous verrons les avantages que celui-ci apporte au sein des bases de données. Nous verrons ensuite à travers un problème de classification les différentes façons d’exécuter du code R ou Python. Enfin, j’ajouterai quelques bonnes pratiques quant à l’utilisation de cette fonctionnalité.
Remarque : N’étant pas Data scientiste, cet article n’est en rien un tutoriel sur le R mais plutôt une présentation de ce qu’il est possible de faire depuis SQL Server ML Services.
1 – Introduction
Dans SQL Server 2016, il est possible d’installer R Services, un module complémentaire à une instance de moteur de base de données utilisé pour l’exécution de code R et des fonctions sur SQL Server.
Dans SQL Server 2017, le R Services a été renommé en SQL Server Machine Learning Services, reflétant l’ajout du langage Python.
Dans SQL Server 2019 (actuellement en Preview), le langage Java est ajouté aux langages R et Python ainsi que le support sur Linux et de haute disponibilité (FCI), le support des données partitionnées…
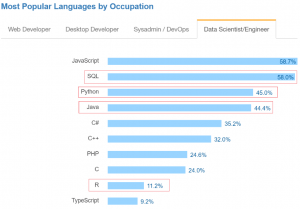
Et ce n’est surement pas au hasard que ces trois langages ont été choisis :
Source : https://insights.stackoverflow.com/survey/2017#technologies-and-occupations
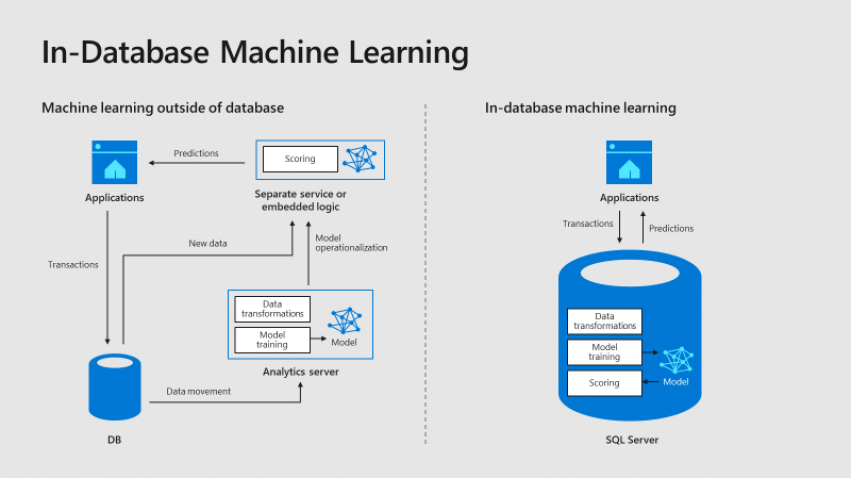
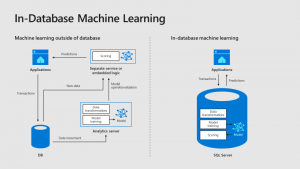
Une tendance dans les architectures actuelles est d’apporter l’intelligence au plus près des données (IoT Edge, …) :
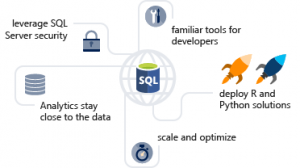
En exécutant un langage de script approuvé au sein d’une infrastructure sécurisée gérée par SQL Server, les administrateurs de base de données peuvent maintenir la sécurité tout en permettant aux scientifiques d’accéder aux données d’entreprise et d’utiliser les ressources des serveurs plutôt que celles de leurs postes de travail.
Le diagramme suivant décrit visuellement les opportunités et les avantages d’une telle architecture :
Exemple de retour client : https://customers.microsoft.com/en-us/story/acxiom-corporation
2 – Démos
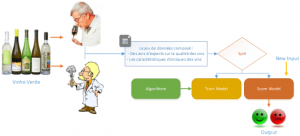
Afin de présenter SQL Server Machine Learning services, prenons une problématique bien réelle et préoccupante : comment prédire la qualité d’un vin sans le goutter !
Pour cela, je vais reprendre les sources de données d’un article que j’avais rédigé en 2014 sur le service Azure Machine Learning : « Predict Wine Quality With Azure ML » https://pulsweb.azurewebsites.net/predict-wine-quality-azureml/. Contrairement à Azure ML qui propose un outil visuel dans le Cloud pour entrainer, créer et déployer un modèle, nous travaillerons dans cet article On Premise dans SQL Server et en mode « Code First ».
Rappel du jeu de données :
– Il a été recueilli par Paulo Cortez, Antonio Cerdeira, Fernando Almeida, Telmo Matos et Jose Reis en 2009 pour la rédaction du livre : Modeling wine preferences by data mining from physicochemical properties. Citation for use: P. Cortez, A. Cerdeira, F. Almeida, T. Matos and J. Reis. Modeling wine preferences by data mining from physicochemical properties. In Decision Support Systems, Elsevier, 47(4):547-553, 2009.
– Les échantillons proviennent de vins blancs et rouges du Portugal : Vinho Verde.
– Les données sont publiques, vous pourriez donc reproduire mes expérimentations : http://www3.dsi.uminho.pt/pcortez/wine/winequality.zip.
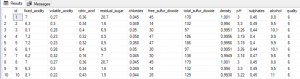
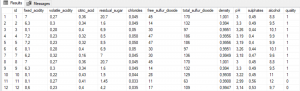
Voici à quoi ressemblent les données :
L’output dans notre exemple sera la qualité. Cette dernière est basée sur des données sensorielles médianes d’au moins 3 évaluations faites par des experts en vin. Chaque expert a classé la qualité du vin entre 0 (très mauvais) et 10 (excellent). Le nombre de vins testés est de 1599 pour les rouges et de 4898 pour les blancs.
Avant de commencer mon expérimentation, revenons sur ce que nous souhaitons faire et obtenir :
2.1 – Architecture 1 : R Cran
Les données sur les vins blancs ayant été chargées en base de données, je peux commencer à les analyser. Voici un exemple de code R exécuté depuis Visual Studio à partir de la version Open Source de R CRAN (3.5.1) :
library(RODBC) # install.packages(« RODBC »)
library(ggplot2) # install.packages(« ggplot2 »)
# Connection
con <- "Driver=SQL Server;Server=MININT-BHOAV8H;Database=WineQuality;Trusted_Connection=yes"
ch <- odbcDriverConnect(connection = con)
# Requete SQL
source.query <- paste("SELECT fixed_acidity, volatile_acidity, citric_acid, residual_sugar, chlorides, free_sulfur_dioxide, total_sulfur_dioxide, density, pH, sulphates, alcohol, quality FROM [dbo].[white];")
wine <- sqlQuery(ch, source.query)
# Affichage des premières lignes
head(wine)
# Description des données
str(wine)
# Graphiques
ggplot(wine, aes(wine$alcohol, wine$pH)) + geom_point(color = "red", size = 0.8) + xlab("Alcohol") + ylab("pH") + ggtitle("Wine Quality")
ggplot(wine, aes(x = quality, fill = quality)) + geom_bar(stat = "count") + geom_text(position = "stack", stat = 'count', aes(label = ..count..), vjust = -0.5) + labs(y = "Num of Observations", x = "Wine Quality") + theme(legend.position = "none")
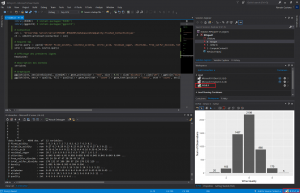
Architecture 1 :
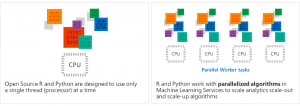
Voyez-vous le problème ? Depuis mon poste de développeur où j’ai installé R CRAN Open Source, je viens d’exécuter une requête sur ma base de données et je viens de rapatrier toutes les colonnes et lignes de ma table « white ».
Avec ce code, toutes les données sont transférées au client et traitées par un seul thread. Voyons donc comment analyser autrement via SQL Server ML Service.
2.2 – Architecture 2 : SQL Server Compute Context
Toujours depuis votre éditeur préféré, en se connectant non plus au R Cran mais au Microsoft R Server installé lors de l’installation de SQL Server :
Exécution du script R suivant :
library(RODBC) # install.packages(« RODBC »)
library(RevoTreeView) # install.packages(« RevoTreeView »)
con <- "Driver=SQL Server;Server=MININT-BHOAV8H;Database=WineQuality;Trusted_Connection=yes"
sqlCompute <- RxInSqlServer(connectionString = con, wait = T, numTasks = 5, consoleOutput = F)
rxSetComputeContext(sqlCompute)
# rxSetComputeContext("local") # if debugging in local !
train <- RxSqlServerData(
connectionString = con,
databaseName = "WineQuality",
sqlQuery = "SELECT * FROM [dbo].[white]",
rowsPerRead = 10000,
stringsAsFactors = T)
# Récupération et affichage des données en local
test <- rxGetInfo(data = train, numRows = 10)
test$data
model <- rxDTree(
quality ~ density + pH + sulphates + alcohol,
data = train,
cp = 0.01)
# Analyse de l'arbre de décision
plot(createTreeView(model))
Résultat :
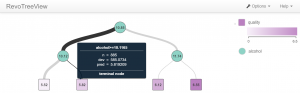
Remarque : Ici les données ne sont pas transférées via le réseau et le code R est exécuté en parallèle côté serveur (au plus proche des données stockées dans SQL Server).
Architecture 2 :
2.3 – Architecture 3 : Procédures stockées
Afin d’exécuter du script R en TSQL, il faut au préalable activer l’exécution de script externe :
EXEC sp_configure ‘external scripts enabled’, 1
RECONFIGURE WITH OVERRIDE
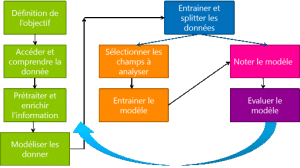
Maintenant rappelons les bases, les principales étapes quant à la construction d’un modèle prédictif :
0. Définition de l’objectif : À partir des caractéristiques d’un vin, nous souhaitons savoir s’il est bon ou mauvais -> Classification binaire.
1. Accéder et comprendre la donnée :
CREATE OR ALTER PROC dbo.[1_Data]
AS BEGIN
SET NOCOUNT ON;
SELECT id, fixed_acidity, volatile_acidity, citric_acid, residual_sugar, chlorides, free_sulfur_dioxide,
total_sulfur_dioxide, density, pH, sulphates, alcohol, quality
FROM dbo.[white];
END;
GO
EXEC dbo.[1_Data];
GO
Résultat :
2. Prétraiter et enrichir l’information :
CREATE OR ALTER PROC dbo.[2_Data_Normalized]
AS BEGIN
SET NOCOUNT ON;
DROP TABLE IF EXISTS dbo.[White_Normalized];
SELECT id, fixed_acidity, volatile_acidity, citric_acid, residual_sugar, chlorides, free_sulfur_dioxide,
total_sulfur_dioxide, density, pH, sulphates, alcohol, IIF(quality<=5,0,1) AS quality
INTO dbo.[White_Normalized]
FROM dbo.[White];
SELECT id, fixed_acidity, volatile_acidity, citric_acid, residual_sugar, chlorides, free_sulfur_dioxide,
total_sulfur_dioxide, density, pH, sulphates, alcohol, quality
FROM dbo.[White_Normalized];
END;
GO
EXEC dbo.[2_Data_Normalized];
GO
Résultat : La colonne « quality » est dorénavant binaire, 0 si le vin est de mauvaise qualité et 1 si le vin est bon.
3. Modéliser les données :
CREATE OR ALTER PROCEDURE dbo.[3_Data_Features] AS
BEGIN
SET NOCOUNT ON;
EXECUTE sp_execute_external_script
@language = N’R’,
@script = N’
library(« reshape2 »)
library(« ggplot2 »)
mainDir <- ''C:\\temp''
dir.create(mainDir, recursive = TRUE, showWarnings = FALSE)
setwd(mainDir);
dest_filename = file.path(mainDir, ''wine_features.jpg'')
jpeg(filename=dest_filename, height = 768, width = 1024, res=150);
numeric_cols <- sapply(wine, is.numeric)
wine.lng <- melt(wine[,numeric_cols], id="quality")
print(ggplot(aes(x=value, group=quality, fill=factor(quality)), data=wine.lng) + geom_density(alpha = 0.2) + facet_wrap(~variable, scales="free")) + theme_classic()
dev.off()',
@input_data_1 = N'SELECT * FROM dbo.[White_Normalized]',
@input_data_1_name = N'wine';
END;
GO
EXEC dbo.[3_Data_Features];
GO
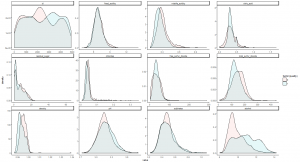
Résultat : la procédure stockée exporte dans un fichier image l’importance des Features pour prédire la qualité du vin. Une courbe superposée ne nous aidera pas à qualifier un vin, il en ressort donc les Features suivantes : citric_acid + total_sulfur_dioxide + density + pH + sulphates + alcohol que j’utiliserai donc pour entrainer mon modèle.
4. Diviser les données (split) :
CREATE OR ALTER PROCEDURE dbo.[4_Split]
AS
BEGIN
SET NOCOUNT ON;
— 75 % Training
DROP TABLE IF EXISTS [dbo].[White_Train];
SELECT * INTO [dbo].[White_Train]
FROM (SELECT * FROM [dbo].[White_Normalized] WHERE (ABS(CAST((BINARY_CHECKSUM(id, NEWID())) as int)) % 100) < 75) a;
-- 25 % Test
DROP TABLE IF EXISTS [dbo].[White_Test];
SELECT * INTO [dbo].[White_Test]
FROM (SELECT * FROM [dbo].[White_Normalized] WHERE id NOT IN (SELECT id FROM [dbo].[White_Train])) a;
SELECT * FROM dbo.White_Train;
SELECT * FROM dbo.White_Test;
END;
GO
EXEC dbo.[4_Split]
GO
Résultat : Sur les 4898 lignes initiales, la table « White_Train » en a 3675 (environ 75 %) et la table « White_Test » en a 1223 (environ 25 %).
5. Entrainement :
CREATE OR ALTER PROCEDURE dbo.[5_Build_Model]
AS BEGIN
DECLARE @inquery nvarchar(max) = N'SELECT * FROM [dbo].[White_Train]';
IF NOT EXISTS (SELECT 1 FROM sys.objects WHERE object_id = OBJECT_ID(N'White_Models') AND type = N'U')
BEGIN
CREATE TABLE [dbo].[White_Models] ([Model] [varbinary](max) NOT NULL, [Date] [DATETIME2](7) NULL);
ALTER TABLE [dbo].[White_Models] ADD CONSTRAINT [DF_Wine_Models_Date] DEFAULT (GETDATE()) FOR [Date];
END
-- DROP TABLE IF EXISTS [dbo].[White_Models];
INSERT INTO [dbo].[White_Models] (Model)
EXEC sp_execute_external_script
@language = N'R',
@script = N'
randomForestObj <- rxDForest(quality ~ citric_acid + total_sulfur_dioxide + density + pH + sulphates + alcohol, InputDataSet)
model <- data.frame(payload = as.raw(serialize(randomForestObj, connection=NULL)))
',
@input_data_1 = @inquery,
@output_data_1_name = N'model';
SELECT * FROM [dbo].[White_Models];
END;
GO
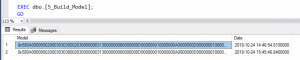
EXEC dbo.[5_Build_Model];
GO
Résultat :
- Entrainement d’arbres de décisions pour prédire la qualité en fonction des Features choisies à partir du jeu de données d’entrainement « White_Train ».
- Sauvegarde du modèle dans une nouvelle table « White_Models » avec l’heure de génération de celui-ci.
6. Noter le modèle (Scoring) :
CREATE OR ALTER PROCEDURE dbo.[6_Score_Model] AS
BEGIN
DECLARE @inquery nvarchar(max) = N’SELECT * FROM [dbo].[White_Test]’;
DECLARE @model varbinary(max) = (SELECT TOP(1) Model FROM dbo.White_Models ORDER BY DATE DESC);
DROP TABLE IF EXISTS [dbo].[White_Predictions];
CREATE TABLE [dbo].[White_Predictions] ([Prediction] [float] NULL, [id] [int] NULL);
INSERT INTO [dbo].[White_Predictions]
EXEC sp_execute_external_script
@language = N’R’,
@script = N’
rfModel <- unserialize(as.raw(model));
OutputDataSet<-rxPredict(rfModel, data = InputDataSet, extraVarsToWrite = c("id"))
',
@input_data_1 = @inquery,
@params = N'@model varbinary(max)',
@model = @model;
SELECT * FROM [dbo].[White_Predictions];
END;
GO
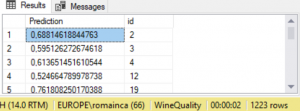
EXEC dbo.[6_Score_Model];
GO
Résultat : sauvegarde des prédictions dans la table « White_Predictions » sur le jeu de données de test « White_Test » (1223 lignes).
7. Évaluer le modèle :
CREATE OR ALTER PROCEDURE dbo.[7_Evaluate_Model] AS
BEGIN
EXEC sp_execute_external_script
@language = N’R’,
@script = N’
suppressMessages(library(« ROCR »))
mainDir <- ''C:\\temp''
dir.create(mainDir, recursive = TRUE, showWarnings = FALSE)
setwd(mainDir);
dest_filename = file.path(mainDir, ''ROC.jpg'')
jpeg(filename=dest_filename, height=1800, width = 1800, res = 300);
pred <- prediction(White_Predictions$Prediction, White_Predictions$Quality)
perf <- performance(pred,"tpr","fpr")
plot(perf)
abline(a=0,b=1)
dev.off()
auc <- performance(pred, "auc")
print(paste0("Area under ROC Curve : ", as.numeric(auc@y.values)))
',
@input_data_1 = N'SELECT b.Prediction, a.Quality FROM [dbo].[White_Test] a INNER JOIN [dbo].[White_Predictions] b ON a.id = b.id',
@input_data_1_name = N'White_Predictions';
SELECT
b.Prediction,
a.Quality
FROM
[dbo].[White_Test] a
INNER JOIN [dbo].[White_Predictions] b ON a.id = b.id
ORDER BY b.Prediction DESC;
END;
GO
EXEC dbo.[7_Evaluate_Model];
GO
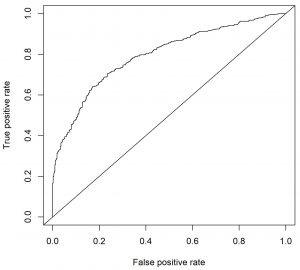
Résultat : Export d’une courbe de ROC
8. Prédiction à la volée :
CREATE OR ALTER PROCEDURE dbo.[8_Prediction]
@citric_acid float, @total_sulfur_dioxide float, @density money, @pH float, @sulphates float, @alcohol float
AS BEGIN
DECLARE @model varbinary(max) = (SELECT TOP 1 Model FROM [dbo].[White_Models]);
EXEC sp_execute_external_script
@language = N’R’,
@script = N’df <- InputDataSet
model <- unserialize(model)
df$Prediction <- rxPredict(model, data = df)
df$Prediction <- as.numeric(df$Prediction)
OutputDataSet <- df',
@input_data_1 = N'SELECT @citric_acid AS citric_acid, @total_sulfur_dioxide AS total_sulfur_dioxide, @density AS density, @pH AS pH, @sulphates AS sulphates, @alcohol AS alcohol;',
@params = N'@model varbinary(max), @citric_acid float, @total_sulfur_dioxide float, @density money, @pH float, @sulphates float, @alcohol float',
@model = @model,
@citric_acid = @citric_acid,
@total_sulfur_dioxide = @total_sulfur_dioxide,
@density = @density,
@pH = @pH,
@sulphates = @sulphates,
@alcohol = @alcohol
WITH RESULT SETS ((citric_acid float, total_sulfur_dioxide float, density money, pH float, sulphates float, alcohol float, PredictedWhiteProb float));
END;
GO
DECLARE @return_value int;
EXEC @return_value = dbo.[8_Prediction]
@citric_acid = 0.45,
@total_sulfur_dioxide = 124,
@density = 0.997,
@pH = 3.2,
@sulphates = 0.46,
@alcohol = 12;
GO
Résultat : Test de deux vins, le premier semble meilleur (l’alcool en serait-il pour quelque chose !)
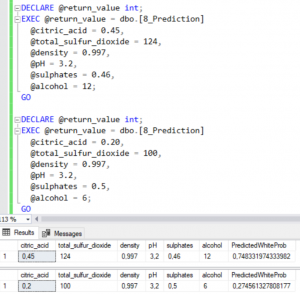
Architecture 3 :
Voici différents objets créés :
3 – Conclusion
Nous avons vu dans cet article différents moyens d’exécuter du R via SQL Server ML Service :
– Depuis un IDE R en changeant de contexte.
– Depuis des procédures TSQL.
Voici quelques bonnes pratiques quant à l’utilisation de SQL Server ML Services :
– Effectuer les transformations de données en TSQL si cela est réalisable plutôt qu’en R ou Python.
– Sauvegarder les modèles en base de données.
– Utiliser des fonctionnalités comme le In Memory, les Columnstore index afin d’améliorer les performances.
– Configurer un pool de ressources pour gérer les ressources externes :
o Create an external resource pool.
o How to create a resource pool for machine learning in SQL Server : https://docs.microsoft.com/en-us/sql/advanced-analytics/administration/how-to-create-a-resource-pool?view=sql-server-2017
– Adapter le montant de la mémoire alloué à l’instance : Server memory configuration options.
– Changer le nombre de compte R créé lors de l’installation (20 par défaut) : Modify the user account pool for machine learning.
– Monitorer l’usage via des rapports SSRS : https://docs.microsoft.com/en-us/sql/advanced-analytics/r/monitor-r-services-using-custom-reports-in-management-studio?view=sql-server-2017
– Installer de nouvelles librairies R, depuis RGui.exe en mode admin :
lib.SQL <- "C:\\Program Files\\Microsoft SQL Server\\MSSQL14.MSSQLSERVER\\R_SERVICES\\library" install.packages("rjson", lib = lib.SQL) Vous trouverez à l’adresse suivant un backup de la base de données ainsi que le projet Visual Studio et les scripts TSQL : https://1drv.ms/f/s!AuHo6XX5MiYSlNojjLWmZGhxBKhEsg